INTRODUCCIÓN
Actualmente, el cáncer de mama es el tipo de cáncer diagnosticado con más frecuencia en las mujeres en todo el mundo.(1) En Cuba, según reportes del registro nacional del cáncer, este representó la segunda causa de muerte por cáncer en las mujeres en 2015.(2,3) Entre todas las variantes histológicas del cáncer de mama, el carcinoma ductal invasivo (CDI) es la más frecuente entre las lesiones invasivas, con alrededor del 80 % de los casos.(4)
El diagnóstico histopatológico preciso de la enfermedad es de vital importancia para guiar su tratamiento efectivo.(5) La detección y delineación de células tumorales en las muestras de tejido es un primer paso en el diagnóstico, seguido de un análisis de estas regiones para determinar el grado y estado del tumor. El análisis visual de dichas muestras en el microscopio es un proceso manual, cualitativo, que consume tiempo y está sujeto a variaciones interobservador, incluso entre patólogos expertos.(6)
Las herramientas de diagnóstico asistido por computadora (CAD, por las siglas del inglés Computer Aided Diagnosis) intentan reducir la carga de los patólogos, automatizando varias tareas de análisis en las imágenes histopatológicas. El objetivo de estos sistemas es complementar el diagnóstico médico mediante la estimación de indicadores cuantitativos que permitan obtener resultados más rápidos, reproducibles y precisos. En este contexto, los algoritmos de aprendizaje automático han demostrado tener alto desempeño en tareas complejas de análisis computarizado de imágenes.(7,8,9) A pesar de esto, el uso de sistemas CAD es escaso en muchos países, incluyendo el nuestro.
Hasta la fecha se ha desarrollado en Cuba un reducido número de sistemas CAD para el análisis computarizado de imágenes histopatológicas digitales. En uno de ellos se desarrolló una herramienta software para el análisis morfométrico de diferentes estructuras celulares en imágenes histopatológicas.(10) La herramienta permitió la estimación de diferentes variables como el área, perímetro, factor de forma y circularidad de las estructuras. Estas métricas fueron utilizadas para la estimación del grado histológico de tumores de CDI mediante un modelo bayesiano de análisis estadístico multivariado; sin embargo, los resultados preliminares mostraron un desempeño de clasificación discreto.(11)
Por otra parte, se desarrolló un sistema para el análisis cuantitativo semiautomático y morfométrico de estructuras celulares en imágenes histopatológicas digitales.(12) Las variables estimadas por este sistema son similares a las descritas en el estudio del software para el análisis morfométrico de diferentes estructuras celulares en imágenes histopatológicas,(10) no obstante se incluyen otras funciones como el conteo de unidades y generación de reportes de datos diagnósticos.
Las herramientas antes mencionadas contienen valiosas funciones para el análisis patológico cuantitativo; sin embargo, no disponen de algoritmos para la detección y delineación automatizada del cáncer de mama en las imágenes. Según el conocimiento de los autores, en Cuba no se cuenta con ningún sistema CAD para la detección/diagnóstico de cáncer de mama en imágenes histopatológicas.
El objetivo de este trabajo fue desarrollar una herramienta software de código abierto que permita detectar y delinear de manera automática las regiones tumorales de CDI en imágenes histopatológicas digitales.
El código fuente de la herramienta se encuentra disponible[a] en la plataforma GitHub.
[a] https://github.com/abrahampm/histobcad
MÉTODOS
En esta sección se describen brevemente los métodos de procesamiento digital de imágenes y aprendizaje automático que soportan la herramienta actual, así como los elementos de desarrollo de software empleados en su diseño.
Método de detección de regiones tumorales
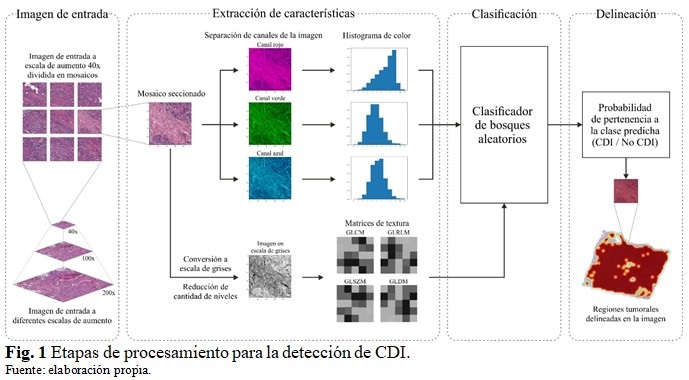
La detección de regiones tumorales de CDI en las imágenes se realiza mediante diferentes bloques de procesamiento. (Fig. 1).
A pesar de que los diferentes tipos de formatos de imágenes histopatológicas contienen múltiples resoluciones, el análisis se realiza a una escala de aumento de 40 ya que esta demostró mejores resultados que otras escalas en un estudio realizado.(13) La imagen de entrada de alta resolución se secciona en mosaicos de 50 50 píxeles para el procesamiento por bloques. Cada mosaico de imagen se procesa de manera independiente en el bloque de extracción de características. Este permite reducir el volumen de información original en la imagen a un conjunto más pequeño de características representativas de color y textura.
Las características de color se calculan mediante el histograma de color normalizado,(14) aplicado a cada canal de la imagen en el espacio de color RGB. Estas brindan información de color de las estructuras celulares (núcleo, estroma, citoplasma) presentes en la imagen, las cuales toman cierta coloración dependiendo del agente de tinción utilizado durante la preparación de las muestras.
Las características de textura permiten obtener información acerca de la distribución espacial y apariencia de las estructuras celulares presentes en las imágenes. Estas se calculan a partir de una serie de matrices descriptivas (GLCM , GLRLM, GLSZM, GLDM)(15,16,17) que caracterizan la dependencia espacial de los niveles de gris en la imagen. Para el cómputo de dichas matrices se convierten los mosaicos de imagen del espacio de color RGB a escala de grises por el método basado en la luminancia.(14) La cantidad de niveles de gris en la imagen influye notablemente en el tamaño de las matrices, por lo que para hacer el cálculo computacionalmente manejable se debe realizar una reducción de los niveles de gris de los mosaicos.(15) El proceso de reducción se realiza dividiendo el rango de intensidades de gris en intervalos discretos de ancho fijo como se define en la ecuación:
El conjunto total de características de color y textura calculadas se proporcionan como entradas al clasificador de bosques aleatorios. Este predice la presencia o no de CDI en cada mosaico de imagen analizado. El entrenamiento del clasificador se realizó utilizando la base de datos pública[b] introducida por Cruz-Roa et al.(9) Esta consiste en 162 imágenes histopatológicas correspondientes a pacientes diagnosticados con CDI. Cada imagen fue anotada manualmente por un patólogo experto y dividida en mosaicos de 50 50 píxeles no superpuestos mediante un muestreo en cuadrícula. Los mosaicos con más del 80 % dentro de la máscara de anotación fueron considerados como positivos (CDI).
La base de datos contiene un total de 277 524 mosaicos de imágenes, 196 454 (71 %) pertenecientes a la clase 0 (No CDI) y 78 768 (29 %) a la clase 1 (CDI). De estos, se seleccionaron para el entrenamiento 123 849 imágenes (45 % del total) realizando un muestreo aleatorio estratificado para preservar las proporciones de las clases presentes en el conjunto original. Para el conjunto de pruebas, se seleccionaron 151 373 imágenes (55 % del total) no empleadas en el entrenamiento, utilizando la misma técnica de muestreo. Las imágenes restantes se descartaron por presentar dimensiones inferiores a 50 50 píxeles.
El clasificador de bosques aleatorios fue entrenado con un total de 100 árboles. Para evitar el sobreajuste del modelo al conjunto de entrenamiento se limitó la cantidad de nodos terminales en cada árbol a 500. Para ser considerado, cada nodo terminal debió apartar al menos 10 ejemplos de entrenamiento a cada rama izquierda y derecha del nodo. Esta configuración de parámetros mostró mejores resultados que otras configuraciones exploradas a través de una búsqueda en malla.
A partir de la probabilidad de pertenencia a la clase predicha por el clasificador para cada mosaico de imagen, se confecciona un mapa de probabilidades que permite finalmente resaltar en colores más cálidos las regiones con alta probabilidad de presencia de CDI en la imagen de entrada.
Desarrollo de herramienta software
El diseño de la herramienta software se realizó en función de una serie de requisitos funcionales y no funcionales descritos a continuación. Entre los requisitos funcionales que debe cumplir la aplicación se encuentran:
- Cargar, visualizar y guardar imágenes histopatológicas digitales.
- Analizar las imágenes y realizar la detección automática de las zonas tumorales de CDI.
- Delinear manual y automáticamente las zonas tumorales detectadas en las imágenes.
- Gestionar y evaluar los datos relativos al diagnóstico del paciente de manera colaborativa.
Por otra parte, la herramienta debe cumplir con requerimientos no funcionales como ser multiplataforma y tener un costo computacional moderado que permita su ejecución en ordenadores con prestaciones limitadas. La aplicación se desarrolló utilizando el lenguaje de programación Python, el cual cumple con el requerimiento multiplataforma, al igual que el marco de trabajo Qt, utilizado para el desarrollo de la interfaz gráfica a través de la integración para Python, PySide2. Se agregó soporte para la lectura de imágenes histopatológicas digitales de distintos formatos a través de la interfaz para Python de la librería OpenSlide. Esta es compatible con varios formatos como Aperio SVS, Leica SCN, Hamamatsu NDPI, entre otros. Debido al gran tamaño en disco y resolución de estas imágenes, la herramienta visualiza solamente la imagen a la escala de aumento en que se realiza el análisis.
La aplicación fue diseñada para que su ejecución se distribuya en diferentes procesos en el sistema operativo anfitrión. Los cálculos intensivos realizados por los algoritmos de procesamiento de imágenes y aprendizaje automático son distribuidos por varios procesos que se ejecutan de manera concurrente en los distintos núcleos disponibles en la CPU del ordenador. Este paralelismo reduce considerablemente los tiempos de ejecución de los algoritmos. Por su parte, la interfaz gráfica se ejecuta por separado en el proceso principal de la aplicación, manteniendo su interactividad mientras se ejecutan las tareas de procesamiento.
Para la evaluación colaborativa se implementó una interfaz de programación de aplicaciones en el lenguaje PHP utilizando el marco de trabajo Laravel. La interfaz se ejecuta en un servidor y permite a los usuarios de la herramienta autenticarse y compartir de manera segura, con otros especialistas, secciones de imágenes histopatológicas, así como datos diagnósticos del paciente para su evaluación colaborativa a distancia. La gestión y almacenamiento de los datos diagnósticos en el servidor se realiza mediante el sistema de gestión de bases de datos MySQL.
[b] Disponible en https://andrewjanowczyk.com/wp-static/ IDC_regular_ps50_idx5.zip
RESULTADOS
La herramienta de código abierto desarrollada se denominó HistoBCAD (del inglés, Histopathological Breast cancer Computer Aided Diagnosis).
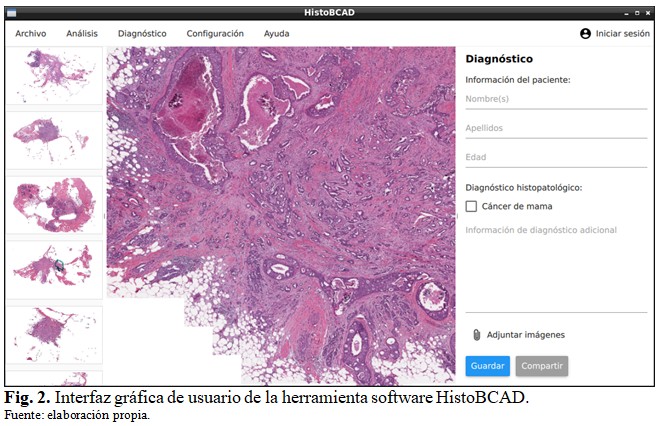
La interfaz gráfica de usuario principal está compuesta por las siguientes partes:
- Barra de menú: contiene una serie de elementos de menú que permiten acceder a las funcionalidades de la aplicación.
- Panel lateral izquierdo: permite previsualizar y acceder rápidamente a todas las imágenes histopatológicas localizadas en el directorio de trabajo actual.
- Panel principal: permite visualizar en detalle la imagen seleccionada, aumentar y disminuir la escala de visualización y desplazar el área visualizada de la imagen.
- Panel lateral derecho: permite gestionar la información de diagnóstico y datos del paciente. (Fig. 2).
Los elementos que integran la barra de menú incluyen las siguientes funciones:
- Menú Archivo: cargar y visualizar una imagen, guardar imagen delineada y cerrar aplicación.
- Menú Análisis: detección automática de regiones tumorales de CDI en la imagen y delineación manual.
- Menú Diagnóstico: crear, editar y compartir datos diagnósticos de los pacientes.
- Menú Configuración: cambiar idioma de la interfaz y ajustes del servidor para el trabajo colaborativo en la aplicación.
- Menú Ayuda: abrir el manual de usuario y mostrar información acerca de la aplicación.
- Elemento de menú Iniciar sesión: gestionar la cuenta de usuario, registro e inicio de sesión en el servidor para el trabajo colaborativo.
El panel lateral derecho contiene varios campos para registrar la información básica del paciente y de diagnóstico. (Fig. 3).
El campo de subtipo histológico permite especificar el tipo de cáncer de mama diagnosticado. En caso de CDI, se habilita otro campo para especificar el grado del tumor (bien diferenciado, moderadamente diferenciado y poco diferenciado). De manera similar, en caso de carcinoma ductal in situ, el subtipo más frecuente entre las variantes in situ de cáncer de mama, se puede especificar su variación histológica (comedo, cribiforme, micropapilar, papilar y sólido). Finalmente, se dispone el campo de información de diagnóstico adicional para introducir en forma de párrafo otros datos y observaciones realizadas durante el diagnóstico.
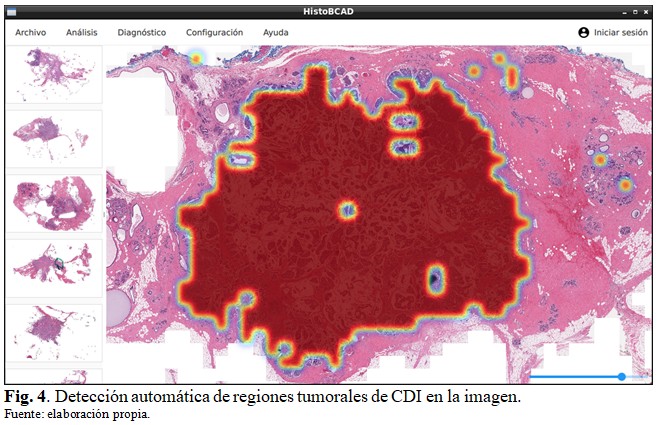
En la figura 4 se muestra la detección automática de regiones tumorales de CDI en una imagen realizada con la herramienta desarrollada. Se resaltan las zonas de alta probabilidad de CDI según la predicción del clasificador realizada para cada mosaico de 50 50 píxeles en la imagen. La detección se realiza en mosaicos para disminuir el costo computacional ya que una detección a nivel de píxel puede resultar excesivamente costosa en ordenadores con recursos computacionales limitados, debido a la alta resolución de las imágenes histopatológicas. (Fig. 4).
El desempeño del algoritmo fue evaluado en el conjunto de pruebas utilizando las métricas de exactitud balanceada y factor F1 como se define en las ecuaciones (2) y (3) respectivamente. Estas métricas son más robustas para la evaluación en conjuntos donde existe desbalance en la cantidad de ejemplos de cada clase.(18)
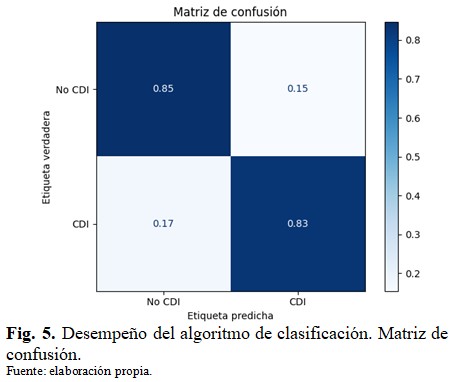
En la figura 5 se muestran los resultados de la evaluación del algoritmo de clasificación para la detección de regiones tumorales de CDI en el conjunto de pruebas mediante la matriz de confusión. La exactitud balanceada alcanzada por el clasificador fue de 84 % y el factor F1 75 %, lo cual representa un resultado competitivo con otros alcanzados por algoritmos de aprendizaje automático convencionales en el tema. (Fig. 5).
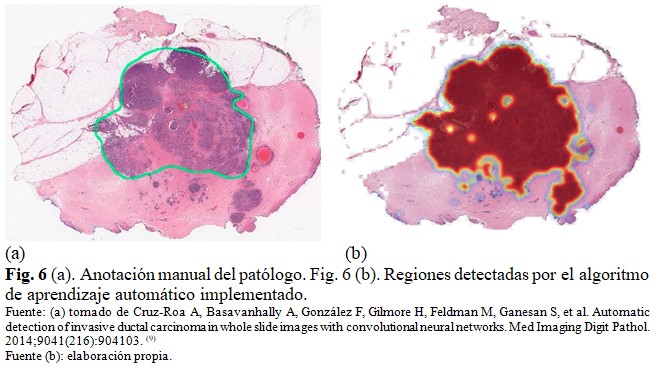
De manera cualitativa, se pudo constatar en las imágenes que, en algunos casos, los falsos positivos o negativos son causados por el bajo nivel de precisión en la anotación manual de referencia realizada en la base de datos original. En la figura 6 se observa un ejemplo, en (a) regiones anotadas manualmente por el patólogo en una imagen original, y (b) regiones detectadas por el algoritmo de aprendizaje automático implementado en la misma imagen reconstruida de la base de datos. (Fig. 6).
Si bien el algoritmo falla detectando incorrectamente algunas regiones, en otros casos el error de clasificación es causado por el bajo nivel de precisión en la delineación manual aproximada realizada por el especialista a una escala de aumento pequeña. La obtención de anotaciones de alta precisión constituye un reto hoy en día en el campo de la histopatología digital debido a la laboriosidad y cantidad de tiempo requerida por dicha tarea.(8)
DISCUSIÓN
HistoBCAD constituye una nueva plataforma de código abierto para el análisis interactivo, reproducible y colaborativo de imágenes histopatológicas digitales en el diagnóstico del cáncer de mama. Esta provee funciones necesarias para la detección automática precisa de regiones tumorales de CDI y demostró ser de ayuda como un primer paso en el proceso de diagnóstico.
El uso de esta herramienta contribuirá a disminuir la carga de trabajo durante el proceso de análisis de grandes volúmenes de imágenes de muestras histopatológicas. Las funcionalidades para la evaluación colaborativa a distancia integradas en la herramienta permiten, además, complementar el diagnóstico con otros criterios aportados por otros especialistas.
En futuras versiones de la aplicación se prevé incorporar nuevos algoritmos de detección y clasificación basados en técnicas de aprendizaje automático supervisado e incremental que permitan asistir otras tareas de análisis durante el proceso de diagnóstico. Se prevé además agregar soporte para la visualización y el análisis de imágenes histopatológicas a diferentes escalas de aumento.
Como trabajo futuro, se propone el estudio y evaluación del método de delineación de regiones tumorales de CDI implementado utilizando otras bases de datos con anotaciones de referencia más precisas.
La herramienta está disponible en: https://github.com/abrahampm/histobcad.
Conflicto de intereses
Los autores declaran que no existen conflictos de interés.
Contribuciones de los autores
Conceptualización: Francisco Perdigón Romero.
Curación de datos: Carlos Abraham Pérez Marrero.
Análisis formal: Carlos Abraham Pérez Marrero.
Adquisición de fondos: Carlos R. Vázquez Seisdedos.
Investigación: Carlos Abraham Pérez Marrero, Francisco Perdigón Romero.
Metodología: Carlos Abraham Pérez Marrero, Carlos R. Vázquez Seisdedos, Francisco Perdigón Romero.
Software: Carlos Abraham Pérez Marrero, Talía Vázquez Romaguera.
Supervisión: Francisco Perdigón Romero.
Redacción – borrador original: Carlos Abraham Pérez Marrero.
Redacción – revisión y edición: Carlos R. Vázquez Seisdedos, Talía Vázquez Romaguera, Alexander Mulet De Los Reyes, Francisco Perdigón Romero.
Financiamiento
El presente artículo fue financiado parcialmente por el proyecto territorial de Monitoreo al adulto mayor en ambientes controlados del Centro de Estudios de Neurociencias, Procesamiento de Imágenes y Señales, Facultad de Ingeniería en Telecomunicaciones, Informática y Biomédica. Universidad de Oriente, Cuba.