INTRODUCCIÓN
La edición de genes manipula la función de estos modificando la secuencia del ácido desoxirribonucleico (DNA) de un organismo. Entre estas tecnologías se encuentran mutagénesis inducida por luz ultravioleta y química, reemplazo genético mediante DNA recombinasa, nucleasas zinc-finger (ZFNs), sistemas nucleasas efectoras semejantes al activador de transcripción (TALEN) y repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas (CRISPR).(1,2,3,4)
CRISPR (del inglés, clustered regularly interspaced short palindromic repeats) fue descubierto por científicos japoneses en 1987.(5) En 2007 se determinó como un complejo sistema inmunitario de los microorganismos para defenderse de la invasión de un gen exógeno; en 2012 se comenzó a emplear en la edición de genes, y tal ha sido su repercusión, que el Premio Nobel en Química de 2020 se otorgó a Emmanuelle Charpentier y Jennifer A. Doudna, por el desarrollo de esta tecnología.(5)
CRISPR/Cas es un método simple, rápido y extremadamente eficiente para la edición de genes; es un poderoso medio para hacer cambios y manipular el genoma de numerosos organismos.(6,7)
La tecnología CRISPR/Cas más empleada es el sistema CRISPR/Cas tipo II que provee a las bacterias de inmunidad adaptativa a virus y plásmidos invasores.(8) Al integrar en su genoma secuencias de DNA idénticas a las de invasores anteriores, las bacterias y arqueas generan una memoria celular de los invasores anteriores. Estas secuencias adquiridas permiten a estos microorganismos reconocer a los invasores virales o plásmidos como no propios, lo que provoca la eliminación de los invasores.
El sistema CRISPR/Cas comprende la nucleasa Cas (del inglés, CRISPR-associated proteins) y dos tipos de ARN, un programable crRNA (CRISPR RNA) y un tracrRNA (trans-activating CRISPR RNA).(2) La proteína Cas es capaz de cortar el ADN del fago invasor en pequeños segmentos, los cuales se integran a la secuencia CRISPR como espaciadores. Posteriormente la secuencia CRISPR se transcribe para generar crRNA y un tracrRNA complementario, los cuales forman una estructura de RNA de doble hélice que recluta proteínas Cas para el clivaje.(2)
Adyacente a la secuencia objetivo del crRNA en el ADN invasor, una secuencia corta denominada motivo adyacente al protoespaciador (PAM, protospacer adjacent motif) desempeña un papel esencial en las etapas de adaptación e interferencia, que el complejo CRISPR/Cas reconoce durante la unión al ADN objetivo. La ausencia de la secuencia PAM puede alterar la afinidad entre Cas y el DNA diana, ya que el reconocimiento específico de la secuencia PAM sirve para discriminar las secuencias diana no propias de las secuencias no diana.(2)
Con el objetivo de optimizar su uso en el laboratorio, el dúplex tracrRNA:crRNA se ha fusionado en una sola molécula de RNA, llamada RNA guía simple (sgRNA) con un programa bioinformático como CRISPR Design Tool (http://tools.genome-engineering.org).(9)
La especificidad es la mayor preocupación en el sistema CRISPR/Cas9, porque Cas9 puede cortar sitios que no son complementarios a la secuencia del sgRNA.(1) Con el objeto de optimizar la predicción de los sgRNA con alta especificidad y reducir la frecuencia relativa de corte de sitios no blanco (off-target), se han desarrollado diversos programas informáticos que sirven como herramientas de apoyo.
CRISPR/Cas9 se aplica en diversos campos de la ciencia como la agricultura y la medicina, ya que puede utilizarse para la edición genómica en microorganismos, plantas, y en células humanas para la erradicación de enfermedades.(4,6,10,11) En esta revisión se discutirán sus principios y aplicaciones en la práctica médica.
DESARROLLO
Métodos
Se realizó una revisión bibliográfica, mediante el uso se Descriptores en Ciencias de Salud (DeCS/MeSH) (https://decs.bvsalud.org/es). Se localizaron “edición de genes”, “edición del genoma”, “sistemas CRISPR-Cas” y “proteína 9 asociada a CRISPR”. Estos se aplicaron a la búsqueda en las bases de datos siguientes: PMC (https://www.ncbi.nlm.nih.gov/pmc); SciELO (https://scielo.org/es); Google académico (https://scholar.google.com.cu); y Cochrane Library (https://www.cochranelibrary.com). En esta última se ubicaron 39 ensayos clínicos sobre la tecnología CRISPR.
Los criterios para la selección de los trabajos: escritos en español o inglés, disponibles a texto completo y sin restricciones en cuanto a los tipos de trabajos y sus fuentes.
Función del locus CRISPR
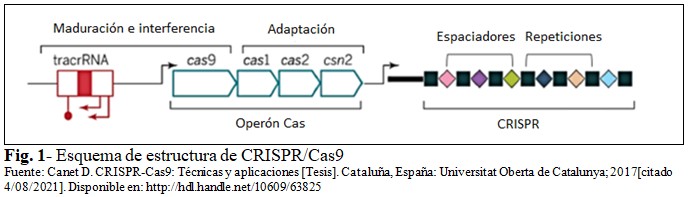
Los sistemas CRISPR/Cas están compuestos por genes organizados en operones y arreglos consistentes en secuencias llamadas espaciadores, interespaciadas por repeticiones idénticas.(1) La función biológica de CRISPR es proteger a los procariotas de la invasión de virus y plásmidos. En la figura 1 aparece una representación esquemática de CRISPR/Cas.(9) (Fig. 1).
Se informan tres tipos de sistemas CRISPR/Cas (I, II y III).(12) En comparación con otros sistemas que necesitan múltiples proteínas Cas, el tipo II solo requiere una única proteína Cas: Cas9.(2) La proteína Cas9 funciona en el reconocimiento y escisión del DNA diana guiado por el sgRNA. En este trabajo se tratará el sistema CRISPR/Cas9 más empleado.
El sgRNA se forma por la unión de dos RNA: crRNA y tracrRNA y forman un complejo con la proteína Cas9.(1) Cada unidad de crRNA contiene una secuencia guía de 20 nucleótidos y una repetición parcial directa, donde la primera dirige a Cas9 a un blanco de 20 nucleótidos a través del apareamiento de bases de Watson-Crick.
Cas9 posee los dominios catalíticos RuvC y HNH, además del dominio de interacción con el PAM.(1) Cas9 realiza cortes de doble hebra (DSB) utilizando los dominios RuvC y HNH. El crRNA y tracRNA se han fusionado para su uso en el laboratorio, dando origen al sgRNA.(1)
El mecanismo general de acción del sistema CRISPR/Cas, independiente de los diferentes tipos, se divide en tres etapas: 1) adaptación, 2) expresión y maduración e 3) interferencia.(13,14) Otros consideran dos fases: adaptación e inhibición.(1)
Durante la fase de adaptación las bacterias o arqueas adquieren una memoria celular del virus o plásmido invasor al integrar secuencias cortas del genoma del virus o del plásmido en el locus CRISPR del genoma de la bacteria o arquea.(8,13,14)
Estos loci CRISPR se identificaron por científicos que trabajaban en la industria de la fermentación. Mediante el análisis genómico comparativo de diferentes cepas de S. thermophiles se determinó un locus altamente variable en el genoma de estas bacterias.(8)
Esta región altamente variable tenía dos características distintas: muchas repeticiones no contiguas, separadas por secuencias variables denominadas protoespaciadores o espaciadores.(8) Luego se descubrió que las bacterias resistentes a los fagos tenían secuencias espaciadoras que coincidían con regiones del genoma de esos fagos.(8) Así, se planteó que regiones cortas del genoma del invasor se integran en loci CRISPR como espaciadores, separados por secuencias repetidas, lo que resulta en una memoria de infecciones anteriores.
Tras la adquisición de los espaciadores, el RNA, denominado crRNA, se genera a partir de los espaciadores en el locus CRISPR y se carga en una proteína Cas.(8) El crRNA dirige a la proteína Cas para que reconozca las secuencias invasoras y corte el ADN del fago o del plásmido entrante.
La expresión y la maduración (biogénesis of crRNA) implica la transcripción de la matriz CRISPR en un ARN precursor-CRISPR (pre-RNA), que se procesa posteriormente para producir crRNA a través de la escisión endonucleotídica y que contiene las secuencias memorizadas del patógeno invasor.(13,14) También se transcribe un segundo RNA procedente de un locus genómico situado aguas arriba del locus CRISPR nombrado tracrRNA.(8)
Cada crRNA contiene un único espaciador (segmento corto de ARN, complementario a la secuencia de ADN del elemento genético extraño) en su extremo 5', y una secuencia repetida CRISPR en su extremo 3'.(13)
De los tres tipos diferentes de sistemas CRISPR-Cas en bacterias y arqueas: I, II y III se tratará el sistema CRISPR tipo II, que ha sido el más comúnmente adaptado para la edición del genoma debido a su simplicidad, el cual requiere solo una proteína Cas, Cas9, y dos componentes de ARN.(8)
El tracrRNA tiene una región complementaria a la región de repetición del locus CRISPR, y se une al pre-RNA recién transcrito, creando un RNA de doble cadena que es cortado por la RNasa III (una enzima que reconoce y corta el RNA de doble cadena) lo que resulta en un complejo crRNA:tracrRNA que contiene solo una secuencia espaciadora.(8) Este complejo de RNA se asocia con una proteína Cas9, creando un complejo de RNP activo.
Durante la etapa de interferencia o inhibición, este complejo efector explora y se dirige a los ácidos nucleicos extraños en la célula.(13) La parte del crRNA del complejo funciona como una guía para reconocer el DNA diana utilizando la secuencia PAM específica aguas arriba o aguas abajo del protoespaciador, mediante el emparejamiento de bases Watson-Crick. El reconocimiento exitoso del DNA diana conduce a su escisión y destrucción por la nucleasa Cas.(13,14)
Una vez que el crRNA:tracrRNA se une a Cas9, esta se activa y puede escindir las secuencias de ácido nucleico invasoras.(8) La búsqueda de secuencias complementarias al crRNA en el DNA invasor se produce mediante la unión de Cas9 a secuencias del genoma viral o del plásmido invasor denominadas PAM.(8)
Diferentes proteínas Cas9 de diferentes especies de bacterias o arqueas reconocen diferentes sitios PAM.(8) Hasta la fecha, la Cas9 de S. pyogenes (SpCas9), que reconoce un PAM 5 0 -NGG-3 0, es la más utilizada para la edición del genoma.(8)
Si el DNA es complementario al sgRNA, se forma un híbrido RNA:DNA, denominado bucle R, y se produce la escisión.(8) El corte del ADN es el resultado de la acción de dos dominios diferentes de la nucleasa Cas9: el dominio HNH corta la cadena complementaria al crRNA y el dominio RuvC corta la cadena que no es complementaria al crRNA.(8,15) Cas9 corta el DNA 3 pares de bases antes de la PAM, lo que resulta en un corte de extremo romo del DNA. El corte del DNA es deletéreo para el plásmido o el virus invasor, lo que resulta en la degradación y la protección contra estos invasores.(8)
Edición de genes en laboratorio
Para editar los genes objetivo, las nucleasas inducen roturas de doble cadena en sitios específicos del genoma, pero antes deben ser incorporadas a las células diana.(3)
Las tecnologías de administración de CRISPR/Cas9 son similares a los métodos de transfección de ácidos nucleicos mediante vectores virales o químicos.(3)
Los métodos para introducir los componentes para la edición del genoma en los organismos o células son: plásmidos de DNA, moléculas de RNA o como complejos de RNP(3,4) Recientemente, han surgido nuevos métodos físicos de transfección debido al desarrollo de la microtecnología y la nanotecnología.(3,16)
A la hora de elegir un formato para la transfección, hay que tener en cuenta muchos factores, como la eficiencia y la duración de la expresión necesaria.(3) La duración de la expresión se ve afectada en gran medida por la velocidad de los procesos biológicos.
Introducción de componentes CRISPR en células
Los vectores de DNA requieren tanto la transcripción como la traducción antes de que pueda producirse la edición del gen.(3) Típicamente, las tasas de transcripción en humanos oscilan entre 6-70 nucleótidos por segundo y las tasas de traducción son aproximadamente cinco aminoácidos por segundo.(3) El DNA entra en el núcleo y se transcribe en el sgRNA y el mRNA que codifica la proteína Cas9.(3) El mRNA de Cas9 se traduce en proteína en el citoplasma, se une al sgRNA y vuelve al núcleo para editar el gen.(3)
La Cas9 se detecta a las cinco horas después de la transfección, alcanza su máximo después de 24-48 horas y a veces tarda semanas antes de que la expresión vuelva a los niveles básicos.(3) Debido a la larga vida media de la Cas9, se prefiere el formato de DNA para los que requieren una mayor duración. Sin embargo, la expresión prolongada del gen Cas9 puede también dar lugar a efectos no deseados fuera del objetivo.(3) Además, Cas9 requiere la transcripción en el núcleo. El DNA del plásmido puede incorporarse al genoma.(3)
El plásmido es más grande que los formatos de mRNA o RNP que se discuten más adelante, lo que puede ser problemático en ciertos métodos de transfección.(3) Además, el DNA del plásmido requiere promotores compatibles para la expresión de sgRNA y Cas9.(3) Aunque el uso de otros formatos es cada vez más frecuente, el DNA plasmídico aún no ha quedado obsoleto debido a su estabilidad y capacidad de expansión.(3)
El formato de mRNA no requiere transcripción y se traduce directamente en el citoplasma antes de que el sgRNA y el Cas9 se localicen en el núcleo.(3) En comparación con el DNA plasmídico, el mRNA es más eficiente en tiempo.(3) La nucleasa Cas9 se expresa una hora después de la transfección, y la expresión máxima se produce en 5-7 horas.(3) Este formato se prefiere para experimentos de duración más corta, lo que también limita los efectos fuera del objetivo.(3)
El formato de RNP es un complejo preformado de Cas9 y sgRNA que no requiere transcripción o traducción.(3) Este formato es el más eficiente y puede funcionar en cuestión de segundos cuando se combina con métodos de transfección como la electroporación.(3) El formato RNP tiene la menor probabilidad de tener efectos fuera del objetivo debido a su velocidad y puede insertar grandes fragmentos de ADN.(3) Sin embargo, los componentes de la RNP pueden ser costosos y son propensos a los contaminantes tóxicos.(3)
Rotura de doble cadena de DNA
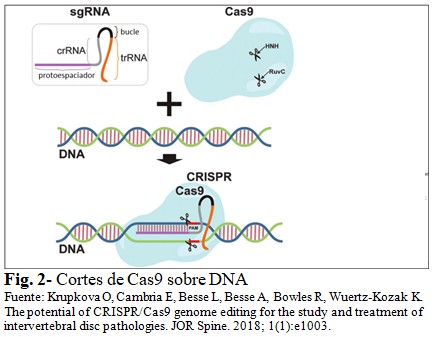
En el nivel más básico, la edición de genes CRISPR/Cas9 tiene dos componentes clave: Cas9, que crea la rotura de la doble cadena de DNA diana (DSB) mediante sus dominios HNH y RuvC; y el sgRNA que "programa" a Cas9 para que corte en sitios específicos.(3,15) (Fig. 2).
Los DSB creados por nucleasas se reparan principalmente por dos procesos de reparación endógena: la unión de extremos no homólogos (NHEJ, non-homologous end joining) y la reparación dirigida por homología (HDR, homology directed repair).(3,17,18,19) El producto resultante de la edición del gen depende del proceso de reparación subyacente.
La NHEJ facilita la reparación de una rotura de doble cadena uniendo los fragmentos de ácido nucleico, en un proceso propenso a errores que produce mutaciones de cambio de marco, codones de terminación prematuros o desintegración sin sentido.(3) La HDR es más precisa al proporcionar una plantilla de DNA que contiene la inserción o corrección deseada, y está flanqueada por brazos homólogos: secuencias de DNA que coinciden con las del sitio de inserción objetivo.(3) Sin embargo, la eficiencia de la HDR es baja en relación con la NHEJ.(3)
En la mayoría de los escenarios, los complejos CRISPR/Cas9 se introducen en el citoplasma de las células objetivo, pero debe atravesar la membrana nuclear, porque la edición genética ocurre en el núcleo.(3) Una secuencia de localización nuclear (NLS, nuclear localization sequence) codificada en el vector del plásmido o en la proteína Cas9 permite que CRISPR/Cas9 entre en el núcleo.(3)
La versión modificada del sistema CRISPR/Cas9 en células de mamíferos comprende la Cas9 optimizada en codones con la NLS que impulsa la compartimentación nuclear en las células de mamíferos.(15) Las NLS que flanquean a Cas9 son más eficientes para dirigir al núcleo.(15)
Además, el crRNA y el tracrRNA se fusionan para crear el sgRNA a través de un bucle diseñado (tetraloop) que conserva las características de los dúplex crRNA-tracrRNA para el reconocimiento de la diana de DNA, el reclutamiento de Cas9 e inducir la rotura de la doble cadena mediada por Cas9.(15)
Aplicaciones de CRISPR/Cas
La tecnología CRISPR/Cas está en franco desarrollo y expansión. Sus perspectivas en el campo de la medicina son amplias, con logrados avances en los últimos tiempos.(20,21,22,23) En Cochrane Library (https://www.cochranelibrary.com) aparecen registrados 39 ensayos clínicos sobre esa novedosa tecnología de edición de genes.
Además de su aplicación in vitro, CRISPR-Cas9 también se emplea para generar modelos animales con vistas a un estudio más efectivo de las enfermedades.(14,24) Por ejemplo, modelos de ratones permiten evaluar el efecto deletéreo de mutaciones en el cáncer, con el uso de mutaciones de pérdida de función en genes supresores tumorales o de ganancia de función en oncogenes.(25,26) Kaushik y colaboradores(27) describen esta tecnología para el tratamiento del cáncer.
La terapia génica puede beneficiarse mucho de la tecnología CRISPR/Cas9.(28) Los esfuerzos para corregir las mutaciones genéticas causantes de enfermedades en humanos, en general tuvieron éxito.(28) Por ejemplo, se empleó para corregir mutaciones causantes de fibrosis quística, β-talasemia, deficiencia de alfa-1 antitripsina, inmunodeficiencia combinada grave y enfermedades neurológicas.(1,13,15,29) La tirosinemia tipo I se corrigió por primera vez en ratones.(17)
La edición de genes de células sanguíneas es atractiva para la cura de enfermedades monogénicas, pero todavía se mantiene experimental. Una revisión sistemática de Maganti y colaboradores,(30) de 3538 estudios preclínicos en animales en vivo, examinó la edición de Hb para corregir la sicklemia (8 estudios), la resistencia inducida al sida (dos estudios) y otras seis enfermedades monogénicas. La persistencia de células con genes editados es el principal desafío.
Se han tratado variaciones genéticas de la diabetes mediante la sustitución de células productoras de insulina en estos pacientes, lo que sugiere que puede emplearse en el tratamiento para la enfermedad diabética no autoinmune.(29) Estos resultados indican que la utilización de las "células del propio paciente" reducía el riesgo de rechazo, además, los pacientes no dependerían de una disponibilidad limitada de donantes externos.
Se ha explorado la edición genética con CRISPR/Cas9 para combatir la infección por HIV.(15,31) Otros estudios sobre virus relevantes como virus de hepatitis B, herpes virus y papiloma virus humano se han realizado utilizando esta tecnología como estrategia antiviral.(32,33)
El sistema CRISPR/Cas se ha utilizado como herramienta antimicrobiana directa.(13) Debido a su alta especificidad, se diseñó una matriz artificial CRISPR para matar selectivamente a las bacterias patógenas dirigiendo su gen de resistencia a los antibióticos y permitir la supervivencia de las cepas no patógenas.
Krupkova y colaboradores(34) describen las perspectivas del empleo de CRISPR/Cas en el tratamiento de las enfermedades del disco intervertebral. También se ha documentado su uso para combatir el mosquito causante de la malaria.(29)
Los usos diagnósticos de la tecnología CRISPR mejoran la eficacia clínica.(6) En 2018 se publicaron artículos sobre su uso en pruebas de diagnóstico para la detección de cáncer en fase temprana o para enfermedades infecciosas.(6) Además, se consideró la innovación más importante para desarrollar un ensayo rápido con vistas a la detección de la tuberculosis.(6)
Es prometedora en la investigación en el área de la agricultura; en particular con CRISPR/Cas9, se han editado exitosamente los genomas de Arabidopsis, tabaco, arroz, trigo, maíz, y tomate.(13) Para ampliar sobre la aplicación en plantas debe revisarse a Razzaq y colaboradores.(4)
Desafíos actuales en el uso de CRISPR/Cas
Aunque CRISPR/Cas9 es el método más popular de edición génica, aun así, se presentan aspectos que siguen siendo explorados y representan desafíos. Se avanza hacia la comprensión de los diferentes subtipos del sistema CRISPR/Cas y el uso de complejos efectores más pequeños (como el Cpf1). En el caso de los vectores virales, preferidos debido a su baja inmunogenicidad, el gran tamaño de los efectores Cas dificulta su empaquetamiento dentro del vector.(1)
Debido a su gran alcance, se produjo la atención mundial sobre las consecuencias que puede tener en el ambiente, la política, la economía y la sociedad.
A pesar de ser útiles en la modificación del genoma y su regulación funcional, los sistemas CRISPR/Cas tienen como principal desafío el efecto fuera del objetivo (no se producen los cortes del DNA en los lugares deseados que no son del todo complementarios a la secuencia guía de sgRNA).(15,17,35) Por ejemplo, la reparación de DSB fuera del objetivo podría originar mutaciones no deseadas.(17)
Desafíos éticos
Sobre CRISPR/Cas9 han surgido varias preocupaciones en relación con la ética y la bioseguridad.(2)
La edición de embriones o la ingeniería de fetos humanos es controvertida entre los científicos.(2,24) Especialmente, algunos países han restringido la tecnología CRISPR/Cas9, o la han prohibido en humanos.(2) Aunque la edición de la línea germinal es útil para entender las enfermedades humanas, las agencias de financiación y organismos reguladores de la investigación son reacios a apoyar esta tecnología por motivos éticos y sociales.(13)
La utilización de CRISPR en embriones corre el riesgo de generar un mosaico genético, además no se puede determinar con exactitud el alcance que tendrá la intervención genómica efectuada.(36) En muchos países no existen normas, o estas investigaciones se consideran como experimentales y no terapéuticas.
Además, los cambios en la línea germinal se transferirán a las generaciones futuras, lo que supone una amenaza potencial para el linaje humano, incluso puede provocar efectos secundarios inesperados a largo plazo.(13,37)
Este avance científico trae inseguridad sobre la posible ausencia de límites en el uso de esta nueva tecnología, ya que puede utilizarse para alterar el genoma, cuyo resultado puede ser nefasto e irreversible.(38) Es necesario suscitar el debate sobre cuál sería el límite de uso en humanos, y hasta dónde ese uso no contribuiría en la erradicación de la diversidad biológica.
Por otra parte, su implementación para eliminar ciertas especies invasivas o alterar determinadas poblaciones salvajes, genera controversia sobre las consecuencias ambientales de la eliminación de especies.(1)
CONCLUSIONES
CRISPR/Cas9 es un sistema de vigilancia inmunitaria que utiliza las bacterias para reconocer el material genético de organismos infecciosos. Con el empleo de Cas y RNA específicos, este sistema escinde los ácidos nucleicos extraños en fragmentos, defendiendo a las bacterias contra los atacantes.
La técnica consiste en el empleo de un sgRNA que se une a secuencias de DNA complementarias, lo que permite a Cas9 romper la doble cadena de DNA diana, luego se reparan esas roturas, lo que facilita la eliminación o modificación de bases específicas en el DNA.
Esta tecnología se ha expandido a un ritmo vertiginoso porque es barata, rápida y fácil de realizar, y ahora se utiliza en casi todos los laboratorios de biología molecular del mundo en un sinfín de organismos para la edición exacta de genomas.
Su aplicación a estudios de genomas, todavía en desarrollo, permitirá una amplia pesquisa de blancos de fenotipos y ayudará a generar modelos experimentales modificados genéticamente para la comprensión y solución de enfermedades en seres humanos.
Aunque los mecanismos regulatorios para esta tecnología están en discusión, es básico abordar este problema desde las perspectivas éticas, sociales, políticas, económicas y ambientales para lograr su aprovechamiento máximo sin prejuicios.
Conflicto de intereses:
Los autores no refieren conflicto de intereses.
Contribución de autoría:
Conceptualización: Rolando Sánchez Artigas,
Análisis formal: Rolando Sánchez Artigas, María Teresa Díaz Armas, Raisa Rodríguez Duque, Pedro Enrique Miguel Soca.
Investigación: Rolando Sánchez Artigas, María Teresa Díaz Armas, Raisa Rodríguez Duque, Pedro Enrique Miguel Soca.
Visualización: María Teresa Díaz Armas, Raisa Rodríguez Duque, Pedro Enrique Miguel Soca.
Supervisión: Rolando Sánchez Artigas,
Redacción – borrador original: Rolando Sánchez Artigas,
Redacción – revisión y edición: Rolando Sánchez Artigas, María Teresa Díaz Armas, Raisa Rodríguez Duque, Pedro Enrique Miguel Soca
Financiamiento:
Ninguno.