INTRODUCCIÓN
Las distrofias musculares de Duchenne (DMD) y de Becker (DMB) son enfermedades neuromusculares progresivas con un patrón de herencia ligado al cromosoma X, causadas por mutaciones (grandes deleciones, duplicaciones y pequeñas mutaciones) en el gen DMD que codifica para la proteína distrofina. Este gen contiene 2,4 Mb y está constituido por 79 exones.1-3 Las grandes deleciones de uno o más exones son las más frecuentes entre las mutaciones del gen y se presentan en el 68,5 % de los casos.1
El gen DMD presenta una alta tasa de mutación y se estima que uno de cada tres casos es causado por una mutación de novo.4,5 El modo de herencia recesivo ligado al cromosoma X, hace que las mujeres portadoras tengan un 50 % de probabilidad de tener hijos varones afectados e hijas portadoras. Esta información es crucial, ya que el conocimiento preciso de la condición de portadora o no portadora genera expectativas y opciones relacionadas con el asesoramiento genético.6-12
En Cuba, la introducción de algunas técnicas de biología molecular como la PCR (reacción en cadena de la polimerasa) -multiplex y el análisis indirecto mediante la construcción de los haplotipos usando repeticiones cortas en tándem de (CA)n (STR por su nombre en inglés Short Tandem Repeats), han hecho posible el análisis de este gen. Ello ha permitido la confirmación diagnóstica en niños con sintomatología clínica de DMD/B, así como la identificación de mujeres por línea materna portadoras de la enfermedad y el diagnóstico prenatal entre las semanas 18-20 del embarazo en aquellas familias con antecedentes de un niño afectado.13,14
El presente estudio tiene como objetivo describir el diagnóstico molecular de distrofia muscular de Duchenne/Becker en una familia sin antecedentes patológicos de la enfermedad.
MÉTODOS
Se realizó un estudio experimental para analizar las deleciones en el gen DMD en un paciente de tres años de edad, con diagnóstico clínico de la enfermedad; los marcadores polimórficos mediante repeticiones cortas en tándem de (CA)n en las mujeres de la familia para identificarlas como portadoras y no portadoras de la enfermedad; y el haplotipo en la familia.
El niño comenzó a ser atendido en la consulta de neurogenética del Instituto de Neurología y Neurocirugía en el año 2012, debido a la presencia de sintomatología clínica de DMD. Luego de la confirmación diagnóstica, la familia asistió a la consulta de asesoramiento genético y se orientó entonces el estudio de las mujeres de la familia, para conocer su condición de portadoras.
Previo consentimiento informado de padres del paciente y sujetos del estudio (seis miembros de la familia), se tomaron 5 ml de muestras de sangre periférica en tubos con EDTA al 5,6%, necesarios para el análisis indirecto. La extracción de ADN genómico se realizó mediante la técnica de precipitación salina.15
En el caso del paciente, la muestra se analizó para identificar las deleciones de 18 exones localizados en los puntos calientes del gen DMD mediante la técnica de PCR-multiplex según los métodos descritos previamente por Chamberlain y Beggs,16,17 con las variantes de tres reacciones independientes para el estudio de nueve exones siguiendo a Chamberlain (mezcla A: 8, 17, 44 y 51; mezcla B: 4, 12, 18 y 45; mezcla C: 44 y 48) y dos reacciones independientes para el estudio de los otros nueve exones siguiendo a Beggs (mezcla 6 exones: promotor, 3, 6, 13, 43, 52; mezcla 3 exones: 47, 50 y 60). El ADN de un sujeto masculino sano sirvió de control positivo, y una reacción sin ADN, como control negativo de amplificación. Posteriormente, 15 µL de los productos de amplificación y 5 µL del marcador de peso molecular pGEM (Promega) fueron resueltos en una electroforesis en gel de agarosa al 3 % y los geles, teñidos con bromuro de etidio. Los resultados fueron visualizados en un transiluminador (Bioblock Scientific).
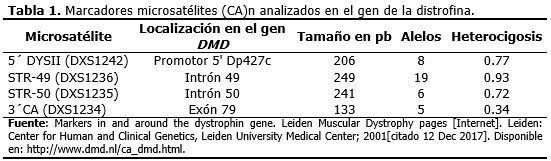
La condición de portadora o no portadora en las mujeres con riesgo se determinó a través del análisis de cuatro microsatélites o STRs situados en el gen DMD (3´CA, STR49, STR50 y 5´ DYS II), disponibles en el laboratorio en ese momento (Tabla 1) y la construcción de los haplotipos, mediante la identificación del alelo mutado por segregación de estos marcadores polimórficos.18,19 El haplotipo de cada STR fue establecido sobre la base de la movilidad electroforética de cada alelo.
Para los STRs se usó una mezcla de PCR a un volumen final de 25 µL, con los reactivos y a las concentraciones siguientes: solución tampón STR 10X a 1X (Promega), 0,04 U/µl de Taq DNA polimerasa (Promega), 1,5 µM de cada primer Eurogentec y 100 ng de ADN genómico. Las amplificaciones se hicieron en un termociclador (Mini CyclerTM MJResearch, PTC-150) en las condiciones específicas para cada STR. Para los STRs 49 y 50, se hicieron a 94 ºC por cuatro minutos, 25 ciclos de 94 ºC por 30 segundos, 62 ºC por 30 segundos, 65 ºC por dos minutos y 65 ºC por siete minutos. Para el STR 3´CA se hicieron a 94 ºC por cinco minutos, 25 ciclos de 94 ºC por 15 segundos, 60 ºC por 30 segundos, 94 ºC por dos minutos y 60 ºC por cinco minutos. Para 5´DYSII se hicieron a 94 ºC por siete minutos, 25 ciclos de 94ºC por 30 segundos, 55 ºC por cuatro minutos, 72 ºC por un minuto y 72 ºC por diez minutos. Para cada estudio de STR se usó un control negativo de amplificación.
Los productos amplificados (15 µl) y 5 µl de un marcador de peso molecular [pGEM (Promega) o Gel Pilot® DNA Molecular Weight Markers (QIAGEN)] se aplicaron sobre geles desnaturalizantes de poliacrilamida al 8 % y se corrieron en electroforesis vertical durante cuatro horas a 560 V, 50 mA. Posteriormente se tiñeron los geles en una solución de bromuro de etidio y se visualizaron en un transiluminador (Bioblock Scientific).
En el año 2017 se adquirió en el laboratorio un fotodocumentador Multi Doc-It™ Imaging Systems UVP acoplado a un trasiluminador Benchtop UV UVP, y acorde a la disponibilidad de muestras de la familia, algunos STRs fueron repetidos y estos geles de poliacrilamida fueron retratados.
Este trabajo ha sido aprobado por el Comité de Ética de la Investigación Científica y por el Consejo Científico del Instituto de Neurología y Neurocirugía.
RESULTADOS
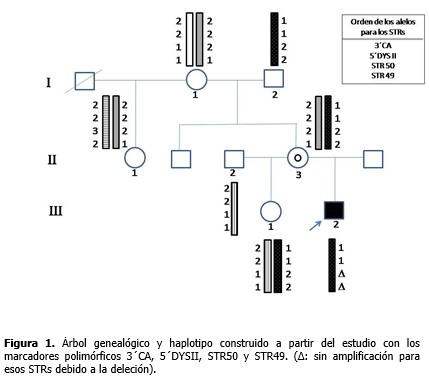
El estudio directo para los 18 exones evaluados del gen DMD permitió identificar en el paciente deleciones de los exones 47 al 52. Con los estudios indirectos se determinó el origen del haplotipo ligado a la enfermedad, gracias a la posibilidad de incluir en el análisis a la abuela y abuelo maternos. Los loci 3´CA, 5´ DYS II, STR 50 y STR 49 mostraron 2, 2, 3 y 2 alelos diferentes, respectivamente para esta familia. (Figura 1).
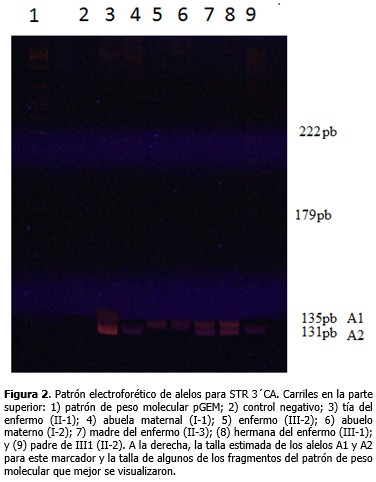
La familia resultó informativa solo para los marcadores 3´CA y 5´DYS II, y con ellos se identificó que el cromosoma X portador de la deleción fue segregado a partir del abuelo materno (I-2), lo cual se observa más detalladamente a través del patrón electroforético de alelos para STR 3´CA. (Figura 2). Del análisis de haplotipos para estos marcadores, claramente se definió que la abuela (I-1) y tía del paciente (II-1) no eran portadoras y, por tanto, no presentaban riesgo de tener hijos afectados por DMD. Para los STRs 49 y 50 no se obtuvo amplificación en el paciente y para el STR 50 la madre del enfermo (II-3) mostró homocigocidad o pérdida de heterocigocidad. (Figura 1).
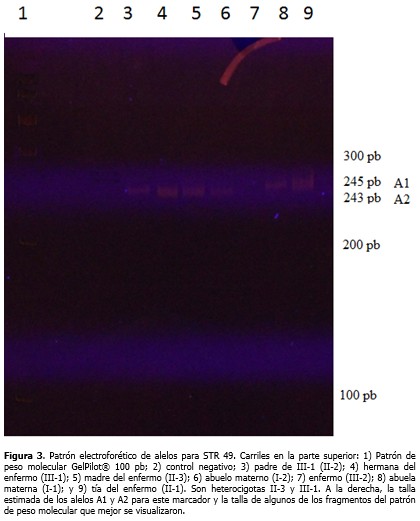
La electroforesis del marcador STR 49 (Figura 3), evidenció no amplificación en carril del enfermo debido a la deleción que incluyó intrón 49 (deleción de los exones 47 al 52). Así mismo, se observó heterocigocidad en II-3, descartándose que la madre fuese una portadora obligada. Su hija (III-1) resultó no portadora dada su heterocigocidad para el locus 49 (Figura 3) y el 50 (Figura 1).
DISCUSIÓN
La no amplificación para los STRs 49 y 50 obtenida en el paciente es un resultado esperado, ya que estos marcadores polimórficos se localizan en la región del gen DMD que se encuentra mutado. La homocigocidad o pérdida de heterocigocidad obtenida para el STR 50 en la madre del enfermo, hizo pensar inicialmente que ella fuera una posible portadora de la deleción, producto de una mutación de novo ocurrida durante la espermatogénesis del abuelo materno. Sin embargo, la heterocigocidad obtenida en ella para el STR 49, descartó esa idea, sin dejar de lado un posible mosaicismo germinal. Los hallazgos de heterocigocidad obtenidos para los locus 49 y 50 en la hermana del niño condujeron a suponer una mutación de novo originada durante la ovogénesis materna de II-3 o durante la embriogénesis de III-2.
El diagnóstico de mujeres portadoras de DMD/DMB ha sido un problema desconcertante por décadas. Las heterocigóticas son, por lo general, clínicamente normales y en casos raros (menos del 5 %) las portadoras muestran síntomas clínicos que pueden variar de moderados a severos. Por lo tanto, en una paciente adulta con debilidad muscular se debe considerar la determinación de su estado de portadora.20
Hoy en día la estrategia diagnóstica más seguida para esta enfermedad consiste en la identificación de deleciones y duplicaciones de exones mediante la técnica de amplificación múltiple de sondas dependientes de ligando, la cual permite examinar los 79 exones que posee el gen DMD. En caso de no hallarse estas mutaciones se procede a la secuenciación de las regiones codificantes y sitios de empalme para la búsqueda de mutaciones puntuales.21-30
Estas mismas técnicas pueden ser utilizadas para la detección de las mutaciones en mujeres en riesgo, pero se recomienda que siempre que sea posible se identifique la mutación en el caso índice o en una portadora obligada de la familia. De otro modo, un resultado negativo es difícil de interpretar, si se considera que estos métodos no son totalmente sensibles y tienen un riesgo residual aproximado al 3 %, aun después de combinadas las técnicas.18 Otras técnicas también han sido utilizadas, tales como: southern blot, hibridación fluorescente in situ y determinación de dosis génica con densitometría o mediante PCR en tiempo real.6,7,21-30
En el laboratorio donde se realizó el estudio, estas técnicas aún no están disponibles. Solo se estudian las deleciones de 18 exones de los 79 exones que posee el gen DMD mediante PCR multiplex y se emplean los polimorfismos (CA)n localizados en este, útiles para la detección de portadoras, mosaicismos germinales y mutaciones de novo.31-33
Según la literatura, las mutaciones de novo de tipo puntual y duplicaciones, surgen preferiblemente durante la espermatogésis, mientras que deleciones ocurren principalmente durante la ovogénisis;20 y en los casos esporádicos son más frecuentes las deleciones distales.34
Grimm y colaboradores20 describen para la DMD/DMB tres situaciones en las que se hereda una mutación en el gen DMD: 1) la madre de un hijo afectado es portadora porque su madre es portadora; 2) la madre de un hijo afectado es portadora como resultado de una mutación de novo que ha surgido en la meiosis durante la espermatogénesis del abuelo, la ovogénesis de la abuela o la ovogénesis de la madre y 3) la madre de un hijo afectado es portadora como resultado de una mutación de novo que ha surgido en la mitosis con la consecuencia de un mosaicismo germinal, durante la espermatogénesis del abuelo, la ovogénesis de la abuela o la ovogénesis de la madre.
En la madre de este niño no se pudo descartar la existencia de un mosaicismo germinal, lo que conllevaría un alto riesgo de ser portadora, aunque pudiera no serlo debido a la ocurrencia de una mutación meiótica de novo en el probando durante la embriogénesis.
Para un caso esporádico donde se confirme una deleción se estima que esa madre tiene la probabilidad de 0,247 de presentar un mosaicismo germinal y de 0,173 de que sea consecuencia de una mutación de novo durante la división meiótica en el enfermo.20
El riesgo de recurrencia (riesgo de un segundo hijo varón afectado) para mujeres no portadoras debido a mosaicismo germinal ha sido estimado entre 14-20 % si el haplotipo de riesgo es trasmitido. Helderman y colaboradores estimaron que en familias con un caso de DMD esporádico de mutación desconocida, el riesgo de recurrencia es más bajo (8,6 %), e incluso, de 4,3 % cuando no existe información sobre el haplotipo de riesgo. Este riesgo es diferente en dependencia del tipo de mutación –deleciones proximales, deleciones distales, duplicaciones y mutaciones puntuales–.35 De cualquier modo, en estos casos, para descartar el riesgo de tener otro hijo afectado se recomienda el diagnóstico prenatal.20
Es significativo recalcar que en los casos DMD/DMB esporádicos con diagnóstico molecular confirmado es importante realizar los estudios por STRs que involucren a varios miembros de la familia, incluyendo los abuelos maternos, pues ello permite identificar correctamente el origen del alelo mutado.
Hasta la fecha se conoce de 419 familias con sospecha clínica de la enfermedad, documentadas en el Libro de Registro Nacional de DMD/DMB, y de ellas solo 121 han solicitado estudios de mujeres portadoras, el 57 % (69/121) correspondiente a casos esporádicos y el 43 % (52/121) a casos familiares. Estas cifras alertan sobre la necesidad de ofrecer un adecuado asesoramiento genético a estas familias, para lograr identificar los riesgos y disminuir la incidencia de la enfermedad.
Puede concluirse que los estudios moleculares realizados permitieron la confirmación diagnóstica de DMD/B en el paciente mediante la identificación de la deleción de los exones 47 al 52, así como la identificación del estado de no portadoras en tres de las cuatro mujeres de la familia estudiadas. Se sugiere la ocurrencia de una mutación de novo. No se pudo excluir un mosaicismo germinal en la madre del paciente. Con estos elementos se pudo brindar un adecuado asesoramiento genético a la familia, al ofrecer una mayor explicación de los riesgos y las opciones reproductivas.